タンパク質‐化学物質相互作用解析システム BioStation
タンパク質-化学物質相互作用マルチスケールシミュレーションシステムBioStationは、タンパク質と化学物質との相互作用を量子化学計算に基づいて解析し、効率的な化合物探索や分子設計を可能にすることを目標に開発を行っているシステムである。非経験的フラグメント分子軌道(FMO)法プログラムABINIT-MP、その計算結果を可視化・解析するBioStation Viewerを中心に、医薬品などの開発の標的となるタンパク質や低分子化合物のデータを収集したデータベースKiBank、これらを統合するBioStation Launcherから構成され、PCクラスタから地球シミュレータまで、幅広い計算環境で利用できる。
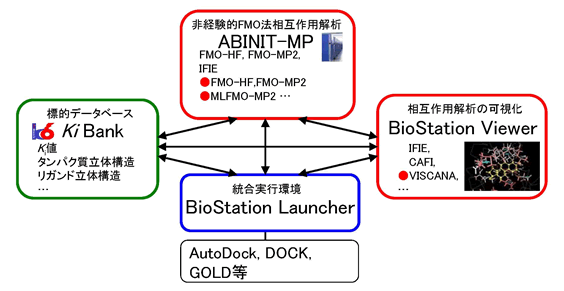
図 1 タンパク質-化学物質相互作用マルチスケールシミュレーションシステム
BioStationシステム構成図
ABINIT-MPはフラグメント分子軌道(FMO)法を実装したプログラムである。タンパク質、DNA、低分子化合物それらの複合体を対象として、分子間相互作用を分子軌道計算に基づいて解析することができる。タンパク質やDNA等の大きな分子をフラグメントと呼ばれる原子団(アミノ酸残基、核酸塩基、リガンド分子など)に分割し、分子軌道計算を並列に処理することで高精度計算を高速に処理している。FMO法の大きな特徴は、フラグメント間の相互作用を計算できることであり、創薬などの分子設計の分野に有用な情報が得られる。
FMO-HF法はHatree-Fock法を基礎とするFMO法であり、電子相関を考慮したFMO-MP2法も利用できる。タンパク質やDNAのようなvan der Walls相互作用や水素結合が重要な系では,FMO-MP2法が必要である。FMO-HF法に対して数倍程度の計算時間でFMO-MP2計算を行うことが可能であり、分散力を考慮した分子間相互作用解析を実用的に行うことができる。
更に、新たに開発された多層化(Multi Layer: ML) FMO-MP2法により、計算資源を計算分子の重要部位に選択的に投入し、この部位のみに対してMP2法を適用することで、解析の精度を落とすことなく計算時間を大幅に短縮することができる。MLFMO-MP2計算を利用した受容体-リガンドドッキング構造の相互作用解析は、医薬品の更なる効率的開発に寄与することが期待される。
BioStation Viewerは、ABINIT-MPによる計算結果を解析・可視化するツールである。分子の立体構造に重ねて、ABINIT-MPの計算結果を表示することができる。フラグメント間相互作用の図示だけでなく、タンパク質-リガンド間相互作用エネルギーのパターンに基づいたリガンドのクラスタリング(VISCANA)を行うことができる。BioStation ViewerはJavaアプリケーションであるため、Java SE とJava 3Dがインストールされていればさまざまな計算機で使用できる。
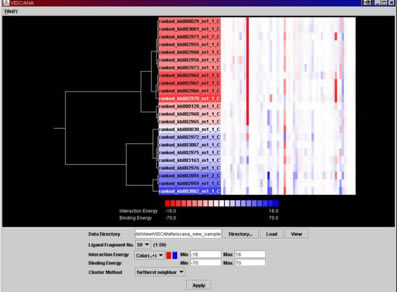
図 2 VISCANAによるリガンドのクラスタリング
KiBankは、タンパク質と低分子化合物との結合親和性実験のデータ(Ki値)を収集したデータベースであり、約16,000件の親和性実験データを収録している。また、リガンド分子とタンパク質に関する基礎データも収録されている。約6,000のリガンド分子および50種のタンパク質の立体構造もあわせて収録されている。これらの立体構造は、欠損原子の補完や水素原子の付加、構造最適化などの処理済であり、直ちに計算に使用できる。KiBankは、ブラウザーを用いて自由に検索できるようにwebデータベースとして公開している。
(http://KiBank.iis.u-tokyo.ac.jp/)
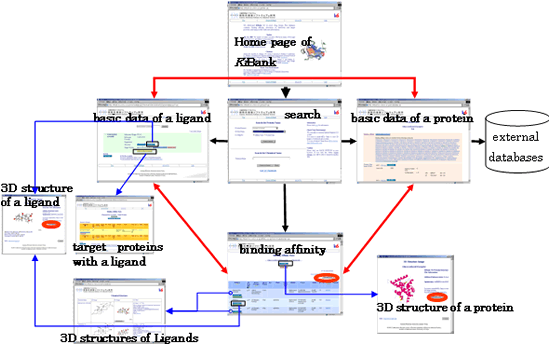
図 3 KiBankの検索サイトマップ
統合環境 BioStation Launcherは、ABINIT-MP、BioStation Viewer、KiBankと市販のドッキンプログラムを統合し、バーチャルリガンドスクリーニングを効率的に行うことが可能となるように設計されており、ABINIT-MPやドッキングプログラムに使いやすいGUIを提供する。スクリーニングを容易に行うことができるようにシンプルなインタフェースを採用している。