- ホーム
- 研究内容
- バイオ分子相互作用シミュレーター
- 研究内容
-
バイオ分子相互作用シミュレーターの研究開発 研究内容
バイオ分子相互作用シミュレーター BioStation
創薬における医薬品の分子設計においては、医薬品の標的となるタンパク質に、より選択的に、より強く結合する化合物をデザインするかが重要な鍵となる。これまでドラッグデザインには、主に古典力場や経験的なスコア関数を用いたドッキングシミュレーションが用いられてきたが、近年、フラグメント分子軌道(Fragment Molecular Orbital; FMO)法[1-8]を用いたタンパク質の量子化学計算に基づいたドラッグデザインへの期待が高まっている。バイオ分子相互作用シミュレータの研究開発(FMO)チームは、BioStation / ABINIT-MPシステムをベースに、FMO法に基づいたバイオ分子相互作用シミュレータの研究開発を行い、創薬におけるプロセスイノベーションに貢献することを目的としている。
バイオ相互作用シミュレータBioStationは、非経験的(ab initio)FMO法を実装したABINIT-MP、その計算結果を可視化し解析するBioStation Viewerから構成され、2013年3月に最新版のABINIT-MP Ver. 6.0およびBioStation Viewer Ver. 15.0を公開した。ABINIT-MPに関しては実行プログラム、技術資料、計算用サンプルデータを、BioStation Viewerに関しては実行プログラム、技術資料、可視化用サンプルデータをダウンロード可能な形で提供している。
ABINIT-MPとBioStation Viewerの具体的な特徴と新機能は以下の通りである。
ABINIT-MPの主な特徴
- エネルギー:多体補正FMO法。計算レベルはHartree-Fock(HF)、2次および3次のMøller-Plesset摂動論(MP2, MP3)に対応
- 構造最適化:HFとMP2で部分構造(フラグメント指定)の最適化が可能
- 解析機能:フラグメント間相互エネルギー解析(IFIE)、自然密度(NBO)電荷
- 並列化:フラットMPIおよびOpenMP/MPI混成
- 対応環境:PCクラスター、超並列スカラー機、ベクトル並列機
- その他:コレスキー分解型高速MP2(Fig.1)、基底関数重ね合わせ誤差(BSSE)の補正
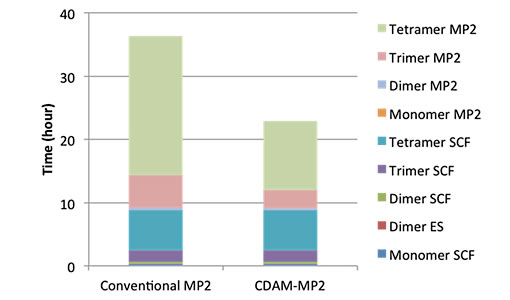
Fig.1 コレスキー分解型高速MP2法によるFMO4計算の高速化ABINIT-MP Ver. 6.0の主な新機能
- FMO4計算(テトラマーフラグメントの計算による多体項補正)(Fig.1)[17,18]
- HF収束性の改良
- OpenMP/MPI混成並列への対応(FMO4-MP2および-MP3)
- Poisson-Boltzmann方程式に基づいた連続溶媒モデルとの連携
- ESPフィッティングによる原子電荷計算
- CAFI法による水素結合ネットワーク解析
- グリッドデータ作成(電子分布および軌道の可視化)機能の統合
- アミノ酸側鎖およびDNA主鎖の自動分割機能
- 大規模計算のための省メモリ化
- IFIEの統計的補正機能(SCIFIE)
BioStation Viewerの主な特徴(Fig.2)
- 入力データ作成:ABINIT-MPの入力ファイルの作成、自動/手動フラグメント分割。
- IFIE解析:IFIEの可視化機能。3次元立体構造表示、網羅的な2次元マップ表示(IFIE map)などが可能
- VISCANA:タンパク質−リガンド相互作用パターン解析によるリガンドのクラスタリング
- 軌道相互作用:水素結合ネットワーク解析(CAFI)、分散相互作用(CH/π, π/π)解析
- CHPI解析:分子座標データからCH/π相互作用を抽出・可視化
- グリッドデータ解析:電子密度、静電ポテンシャル、分子軌道、電場ベクトルの表示
- 対応環境:Windows
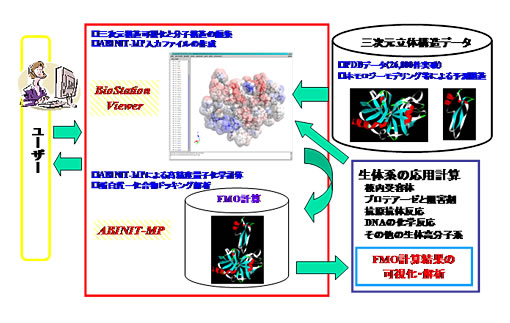
Fig.2 BioStationシステム利用のイメージBioStation Viewer Ver. 15.0の主な新機能
- 自動フラグメント分割機能の拡張(アミノ酸側鎖、DNA主鎖、SiO2結晶)
- 多体FMO計算の入力データ作成と結果の表示
- 超分子計算結果の可視化
- 大規模な解析結果表示のための省メモリ化
【参考文献】
- D. G. Fedorov, K. Kitaura, "Theoretical development of the fragment molecular orbital (FMO) method", in Modern methods for theoretical physical chemistry of biopolymers, E. B. Starikov, J. P. Lewis, S. Tanaka, Eds., Elsevier (2006) pp. 3-38.
- T. Nakano, Y. Mochizuki, K. Fukuzawa, S. Amari, S. Tanaka, "Developments and applications of ABINIT-MP software based on the fragment molecular orbital method", in Modern methods for theoretical physical chemistry of biopolymers, E. B. Starikov, J. P. Lewis, S. Tanaka, Eds., Elsevier (2006) pp. 39-52.
- D. G. Fedorov, K. Kitaura, "Extending the Power of Quantum Chemistry to Large Systems with the Fragment Molecular Orbital Method", J. Phys. Chem. A, 111, 6904-6914 (2007).
- 中野達也, 望月祐志, 甘利真司,小林将人,福澤薫,田中成典, "フラグメント分子軌道法に基づいた生体巨大分子の電子状態計算の現状と今後の展望", J. Comput. Chem. Jpn. 6, 173-184 (2007). (http://www.sccj.net/publications/JCCJ/v6n3/a06/document.pdf)
- 福澤薫, 中野達也, 加藤昭史, 望月祐志, 田中成典, "フラグメント分子軌道法による生体高分子の応用計算", J. Comput. Chem. Jpn. 6, 185-198 (2007). (http://www.sccj.net/publications/JCCJ/v6n3/a07/document.pdf)
- 北浦和夫, 計算化学研究会2008.05.22 GAMESS/FMOワークショップ資料 (http://www.cbi.or.jp/cbi/jigyou/FMO_gaiyo.pdf)
- 佐藤文俊,中野達也,望月祐志 編著,"プログラムで実践する生体分子量子化学計算", (森北出版,東京,2008).
- "The Fragment Molecular Orbital Method: Practical Applications to Large Molecular Systems", D.G. Fedorov, K. Kitaura, Eds., (CRC Press, Boca Raton, 2009).
- Y. Mochizuki, K. Yamashita, K. Fukuzawa, K. Takematsu, H. Watanabe, N. Taguchi, Y. Okiyama, M. Tsuboi, T. Nakano, S. Tanaka, "Large-scale FMO-MP3 calculations on the surface proteins of influenza virus, hemagglutinin (HA) and neuraminidase (NA)", Chemical Physics Letters, 493, 346-352 (2010).
- I. Kurisaki, K. Fukuzawa, T. Nakano, Y. Mochizuki, H. Watanabe, and S. Tanaka, " Fragment molecular orbital (FMO) study on stabilization mechanism of neurooncological ventral antigen (NOVA)-RNA complex system", J. Mol. Struct. (THEOCHEM), 962, 45-55 (2010).
- Y. Mochizuki, T. Nakano, Y. Komeiji, Y. Okiyama, H. Yoshikawa, H. Yamataka, " Fragment molecular orbital-based molecular dynamics (FMO-MD) method with MP2 gradient", Chemical Physics Letters, 504, 95-99 (2011).
- N. Taguchi, Y. Mochizuki, T. Nakano, "Fragment molecular orbital calculations for excitation energies of blue- and yellow-fluorescent proteins", Chemical Physics Letters, 504, 76-82 (2011).
- Y. Umezawa, M. Nishio, "CH/π Interactions as Demonstrated in the Crystal structure of Guanine-nucleotide Binding Proteins, Src homology-2 Domains and Human Growth Hormone in Complex with their Specific Ligands", Bioorganic & Medicinal Chemistry Volume 6, Issue 4, Pages 493-504 (1998).
- M. Nishio, M., Hirota, Y. Umezawa, "The CH/π Interaction Evidence, Nature, and Consequences", Wiley-VCH, New York (1998).
- 西尾元宏「新版 有機化学のための分子間力入門」講談社2008.
- Y. Okiyama, K. Fukuzawa, H. Yamada, Y. Mochizuki, T. Nakano, S. Tanaka, "Counterpoise-corrected interaction energy analysis based on the fragment molecular orbital scheme", Chem. Phys. Lett., 509, 67-71 (2011).
- T. Nakano, Y. Mochizuki, K. Yamashita, C. Watanabe, K. Fukuzawa, K. Segawa, Y. Okiyama, T. Tsukamoto, S. Tanaka, "Development of the four-body corrected fragment molecular orbital (FMO4) method", Chem. Phys. Lett. 523, 128-133 (2012).
- C. Watanabe, K. Fukuzawa, Y. Okiyama, T. Tsukamoto, A. Kato, S. Tanaka, Y. Mochizuki, T. Nakano, "Three- and four-body corrected fragment molecular orbital calculations with a novel subdividing fragmentation method applicable to structure-based drug design", Journal of Molecular Graphics and Modelling, 41, 31-42 (2013).
Copyright © Collaborative Center for Research on Innovative Simulation Software,The University of Tokyo

